分子間相互作用解析システムによるタンパク質相互作用の評価
Estimation of protein-protein interactions using Surface Plasmon Resonance
(担当:谷口 博昭)
(Member in charge: Hiroaki Taniguchi)
【概要】
Summary
特定の生命現象あるいは組織・細胞等を対象とした遺伝子発現解析を網羅的に行うことにより、当該対象物に特異性のある発現を示す遺伝子や特定の形質との相関性を認めた遺伝子を見出し、さらに次の段階としてその遺伝子産物の機能をin vitroやin vivoの複数の実験系で検証することが一般的である。
in vitroの機能解析の一端として、事前の研究により同定した、研究対象との関連性が深いと想定される遺伝子産物と「相互作用するタンパク質」を同定することにより、当該する遺伝子産物の機能を推定することが可能である。さらに、遺伝子産物と相互作用するタンパク質の結合が立証された場合、それらの結合を阻害する可能性がある低分子化合物の評価が可能であり、新規創薬へ繋げることが可能となる。
本支援では、表面プラズモン共鳴分析法による分子間相互作用解析システム(Biacore X100 plus)を用いて、被支援者が研究対象とする遺伝子産物と相互作用するタンパク質との相互作用を結合定数および解離定数の算定に基づいて同定する。
In general, after comprehensive gene expression analysis with cancer tissues and cells has done for a particular biological phenomenon, particular genes are found to be related to special phenotype. Next step, functions of gene products – proteins – are validated by in vitro or in vivo analysis.
The functions of target proteins are speculated from data of protein-protein interactions (PPIs) related to them as part of in vitro analysis. It is possible to estimate low-molecular-weight compounds as inhibitors for PPI after confirmation of binding between target proteins and other proteins using Surface Plasmon Resonance (SPR). Consequentially, estimation of compounds lead to drug development.
SPR is one of the most commonly used techniques to estimate PPIs. The advantage of SPR is able to measure the binding affinities and association/dissociation kinetics of complexes in real time, in a label-free environment, and using relatively small quantities of materials.
In our support, we provide the data of binding affinities and association/dissociation kinetics for PPIs of proteins provided from researchers.
【材料・方法】
Materials and Methods
まず、研究対象とする遺伝子産物(リガンド)のセンサーチップ上への固定化条件を検討する。センサーチップ上にリガンドを固定化する際、結果が比較的安定に得られる固定化の方法である、GST融合遺伝子産物(リガンド)のGST tagを介した固定(センサーチップ上の抗GST抗体で捕捉)を用いる。もしくは、我々の提供するFLAG-GST-目的蛋白質を発現するベクターを被支援者に提供する。被支援者に高純度のGST融合タンパク質(リガンド)、または、FLAG-GST-融合タンパク質(リガンド)、及び、相互作用するタンパク質候補(アナライト)をご準備頂く(図.1)。また、GST tagを介した固定化を用いるため、アナライトにGST tagタンパク質は使用できない。
リガンドとアナライトの情報より、センサーチップ上へのリガンドの固定化量を算出する。具体的には、最大結合量 (Rmax) [= (アナライト分子量/リガンド分子量) X (リガンドの固定化量) X (結合比率)] の値が最低で20 – 100RUが必要となる。例えば、Rmax値 50を確保する場合、分子量比率より必要とされるリガンドの固定化量を算出し、リガンドの固定化条件を検討する。
次に、相互作用するタンパク質候補(アナライト)を流路に流し、分子間相互作用解析システム(Biacore X100 plus)により、リガンドとの結合・解離を確認する(結合定数・解離定数の算定)。
The method is based on the immobilization of the binding partner (the ligand) as GST-fusion recombinant protein via GST antibody, on a sensor chip surface. We can also provide an expression vector (as a backbone) of FLAG-GST-fusion proteins to express ligand proteins. Immobilization is followed by the injection of the other partner (the analyte), except for GST tagged protein, over the surface containing the ligand. The binding is monitored by subsequent changes in the refractive index of the medium close to the sensor surface upon injection of the analyte(Fig. 1). Protocols are constituted by the immobilization of proteins with GST capture method, analyte-binding analysis, affinity/kinetic measurements, and data analysis using Biacore X100 plus.
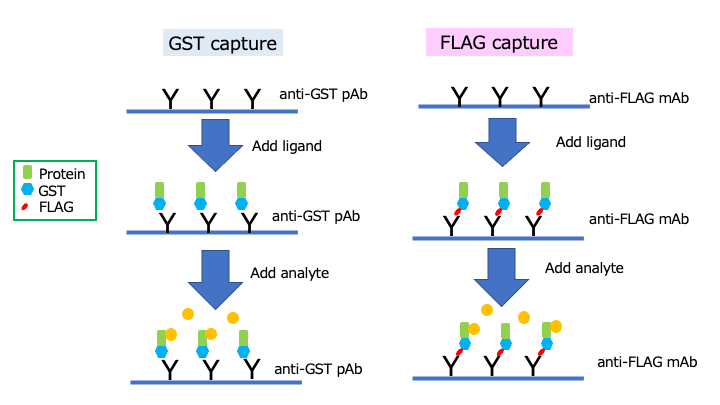
Fig. 1.(図.1) GST capture method and FLAG capture method
【応募条件】
Requirement of the support
- 本支援項目は共同研究ベースで実施させて頂きます。
- 被検タンパク質は被支援者にご準備頂きます。
- 固相側は基本的にGST融合タンパク質、もしくはFLAG-GST融合蛋白質とします。
- We perform SPR analysis as collaboration study.
- Please prepare recombinant proteins by researchers.
- The ligand has to be GST-fusion or FLAG-GST-fusion recombinant proteins due to using GST capture method.
【参考文献】
Reference
- Moriya C, Taniguchi H, Nagatoishi S, Igarashi H, Tsumoto K, Imai K. PRDM14 directly interacts with heat shock proteins HSP90α and GRP78. Cancer Sci. 109: 373-383 (2017). https://doi.org/10.1111/cas.13458

